9 Árboles de decisión
En las secciones anteriores, vimos cómo generalmente para utilizar métodos estructurados como regresión (lineal o logística), es necesario hacer trabajo considerable para crear variables de entrada apropiadas. En caso contrario, muchas veces sufrimos de problemas de sesgo (junto posiblemente varianza). Hemos utilizado por ejemplo:
- Transformaciones no lineales y expansión no lineal de entradas
- Categorización de variables ordinales (escalas)
- Distintos tratamientos de variables categóricas
- Introducción de interacciones entre variables de entrada
- Transformaciones y filtrado para mejorar desempeño cuando hay valores atípicos en las entradas
- Existen muchos otros más.
Notamos también que este proceso de expansión de entradas produce modelos grandes (dimensión alta) que muchas veces se benefician controlando varianza usando regularización o métodos de selección de variables.
Quisiéramos ahora estudiar métodos más automáticos para hacer este trabajo de ingeniería de entradas, evitando en lo posible los problemas que hemos considerado de dimensión alta. Dos familias útiles de métodos son:
- Árboles de decisión y métodos de agregación de árboles (como bosques aleatorios o boosting).
- Redes neuronales.
El enfoque de estos dos métodos es distinto, pero ambos pretenden “crear” las variables derivadas necesarias para tener buen desempeño predictivo. Primero consideramos un ejemplo más de cómo este proceso de ingeniería de entradas es importante al usar modelos como regresión
9.1 Ejemplo: cancelación de reservaciones
Consideramos el problema de predecir qué reservaciones de un hotel son canceladas. Este problema es parte importante de la planeación de un hotel pues es parte de la construcción de previsiones necesarias para la administración del inventario (cuartos por noche). Estos datos provienen de este artículo y este que también explican cosas importantes del problema y posibles soluciones.
hoteles <- read_csv("./datos/muestra_hoteles.csv")
set.seed(3343)
particion_hoteles <- validation_split(hoteles, 0.9)
particion_hoteles[[1]]## [[1]]
## <Training/Validation/Total>
## <82377/9154/91531>Con esta partición tenemos casi 10 mil casos para validar y alrededor de 80 mil para entrenar. Consideramos primero un modelo simple:
entrena_tbl <- training(particion_hoteles$splits[[1]])
receta_hoteles <- recipe(is_canceled ~ hotel + lead_time + country +
is_repeated_guest + deposit_type +
customer_type +
distribution_channel + market_segment, entrena_tbl) |>
step_other(country, threshold = 0.005) |>
step_dummy(all_nominal_predictors()) ## [1] "lead_time" "is_repeated_guest"
## [3] "is_canceled" "hotel_Resort.Hotel"
## [5] "country_BEL" "country_BRA"
## [7] "country_CHE" "country_CHN"
## [9] "country_CN" "country_DEU"
## [11] "country_ESP" "country_FRA"
## [13] "country_GBR" "country_IRL"
## [15] "country_ISR" "country_ITA"
## [17] "country_NLD" "country_POL"
## [19] "country_PRT" "country_SWE"
## [21] "country_USA" "country_other"
## [23] "deposit_type_Non.Refund" "deposit_type_Refundable"
## [25] "customer_type_Group" "customer_type_Transient"
## [27] "customer_type_Transient.Party" "distribution_channel_Direct"
## [29] "distribution_channel_GDS" "distribution_channel_TA.TO"
## [31] "distribution_channel_Undefined" "market_segment_Complementary"
## [33] "market_segment_Corporate" "market_segment_Direct"
## [35] "market_segment_Groups" "market_segment_Offline.TA.TO"
## [37] "market_segment_Online.TA" "market_segment_Undefined"
flujo_hoteles <- workflow() |> add_recipe(receta_hoteles) |>
add_model(logistic_reg(engine = "glmnet", mixture = 0.1, penalty = 0.0001))
fit_resamples(flujo_hoteles, particion_hoteles, metrics = metric_set(roc_auc, mn_log_loss)) |>
collect_metrics()## # A tibble: 2 × 6
## .metric .estimator mean n std_err .config
## <chr> <chr> <dbl> <int> <dbl> <chr>
## 1 mn_log_loss binary 0.432 1 NA Preprocessor1_Model1
## 2 roc_auc binary 0.847 1 NA Preprocessor1_Model1Si hacemos un poco de investigación descubrimos por ejemplo que los que pagaron sin posibilidad de reembolso tienen una tasa de cancelación muy alta:
entrena_tbl |> count(deposit_type, is_canceled) |>
group_by(deposit_type) |>
mutate(prop = n / sum(n))## # A tibble: 6 × 4
## # Groups: deposit_type [3]
## deposit_type is_canceled n prop
## <chr> <chr> <int> <dbl>
## 1 No Deposit cancelado 18953 0.265
## 2 No Deposit no_cancelado 52686 0.735
## 3 Non Refund cancelado 10531 0.992
## 4 Non Refund no_cancelado 83 0.00782
## 5 Refundable cancelado 9 0.0726
## 6 Refundable no_cancelado 115 0.927Y según el paper, esto se debe principalmente a que son reservaciones falsas (con tarjeta de crédito falsa) que se utilizan para hacer solicitudes de visa en Portugal. Podemos entonces poner una interacción de este tipo de depósitos con Portugal.
Adicinalmente, investigando cómo se relaciones la antelación (lead time) con la variable respuesta, decidimos convertirla a logaritmo, y ponerla en interacción con cada país. Tenemos ahora la siguiente receta más complicada
receta_hoteles_2 <- recipe(is_canceled ~ hotel + lead_time + country +
is_repeated_guest + deposit_type +
customer_type +
distribution_channel + market_segment, entrena_tbl) |>
step_other(country, threshold = 0.005) |>
step_log(lead_time, offset = 1) |>
step_dummy(all_nominal_predictors()) |>
step_interact(~ starts_with("lead_time"):starts_with("country")) |>
step_interact(~ starts_with("lead_time"):starts_with("hotel")) |>
step_interact(~ country_PRT:deposit_type_Non.Refund) |>
step_interact(~ starts_with("market_segment"):starts_with("customer_type"):starts_with("distribution"))que sin embargo da mejores resultados:
flujo_hoteles <- workflow() |> add_recipe(receta_hoteles_2) |>
add_model(logistic_reg(engine = "glmnet", mixture = 0.1, penalty = 0.0001))
fit_resamples(flujo_hoteles, particion_hoteles, metrics = metric_set(roc_auc, mn_log_loss)) |>
collect_metrics()## # A tibble: 2 × 6
## .metric .estimator mean n std_err .config
## <chr> <chr> <dbl> <int> <dbl> <chr>
## 1 mn_log_loss binary 0.409 1 NA Preprocessor1_Model1
## 2 roc_auc binary 0.869 1 NA Preprocessor1_Model1Construir las transformaciones e interacciones apropiadas requiere trabajo considerable de exploración y entendimiento de los datos. Veremos cómo es posible incluir interacciones apropiadas en el proceso de ajuste.
9.2 Árboles de decisión
Antes de expicar con detalle el funcionamiento y construcción de árboles para clasificación, comenzamos por ver el desempeño de un árbol de decisión usando tidy models para problema de reservas, usando la receta simple original sin preprocesamiento.
Ejemplo: cancelación de hoteles
modelo_arbol <- decision_tree(tree_depth = 8, cost_complexity = 0.0001) |>
set_mode("classification") |>
set_args(model = TRUE)
flujo_hoteles <- workflow() |> add_recipe(receta_hoteles) |>
add_model(modelo_arbol)
fit_resamples(flujo_hoteles, particion_hoteles,
metrics = metric_set(roc_auc, mn_log_loss)) |>
collect_metrics()## # A tibble: 2 × 6
## .metric .estimator mean n std_err .config
## <chr> <chr> <dbl> <int> <dbl> <chr>
## 1 mn_log_loss binary 0.388 1 NA Preprocessor1_Model1
## 2 roc_auc binary 0.877 1 NA Preprocessor1_Model1Y vemos que superamos fácilmente en desempeño nuestros intentos anteriores. Examinamos el árbol ajustado:
library(rpart.plot)
flujo_ajustado <- fit(flujo_hoteles, entrena_tbl)
arbol <- flujo_ajustado |> extract_fit_engine()
prp(arbol, type = 4, extra = 4)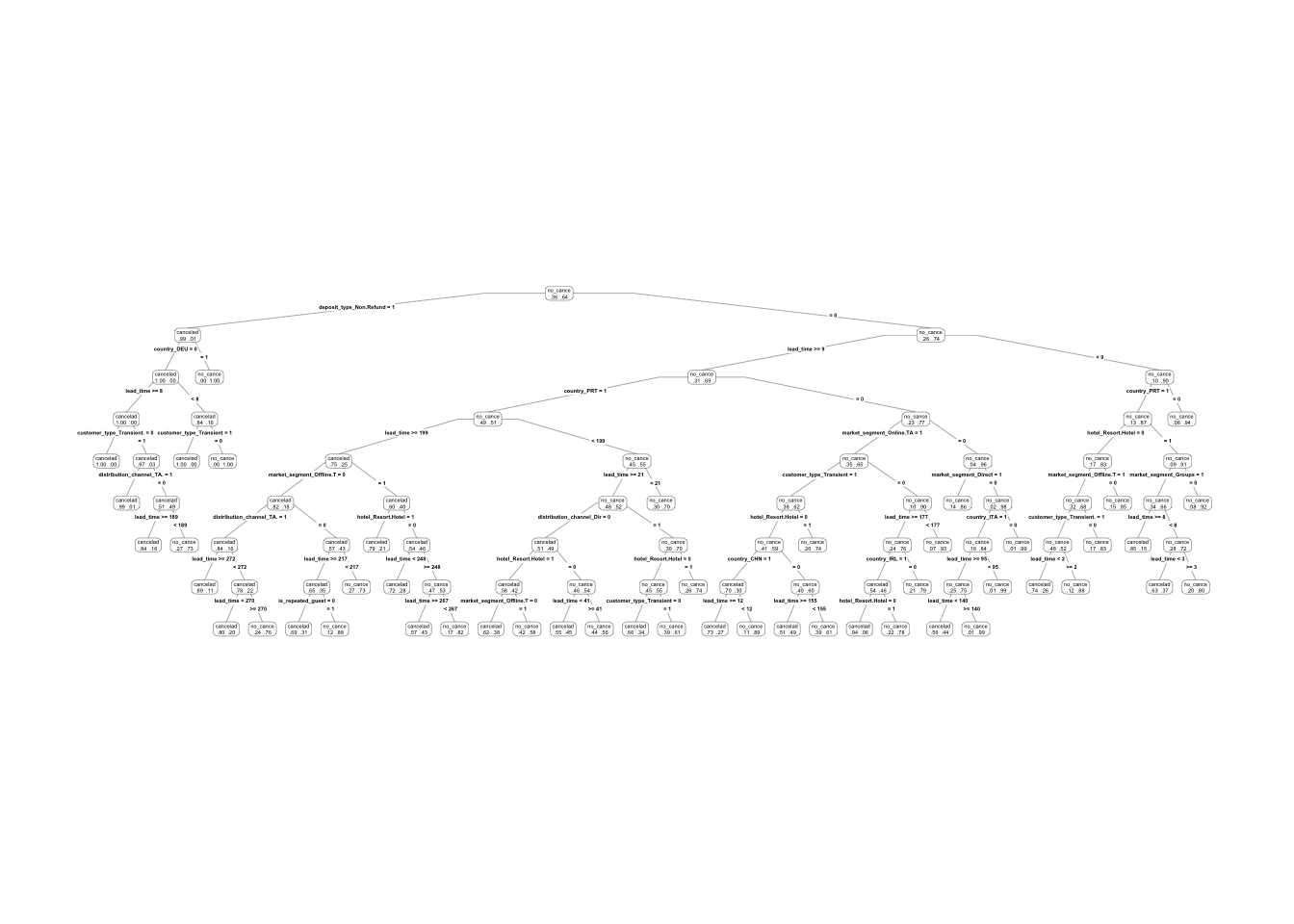 Este árbol es considerablemente grande, pero podemos examinar comenzando por los
primeros cortes:
Este árbol es considerablemente grande, pero podemos examinar comenzando por los
primeros cortes: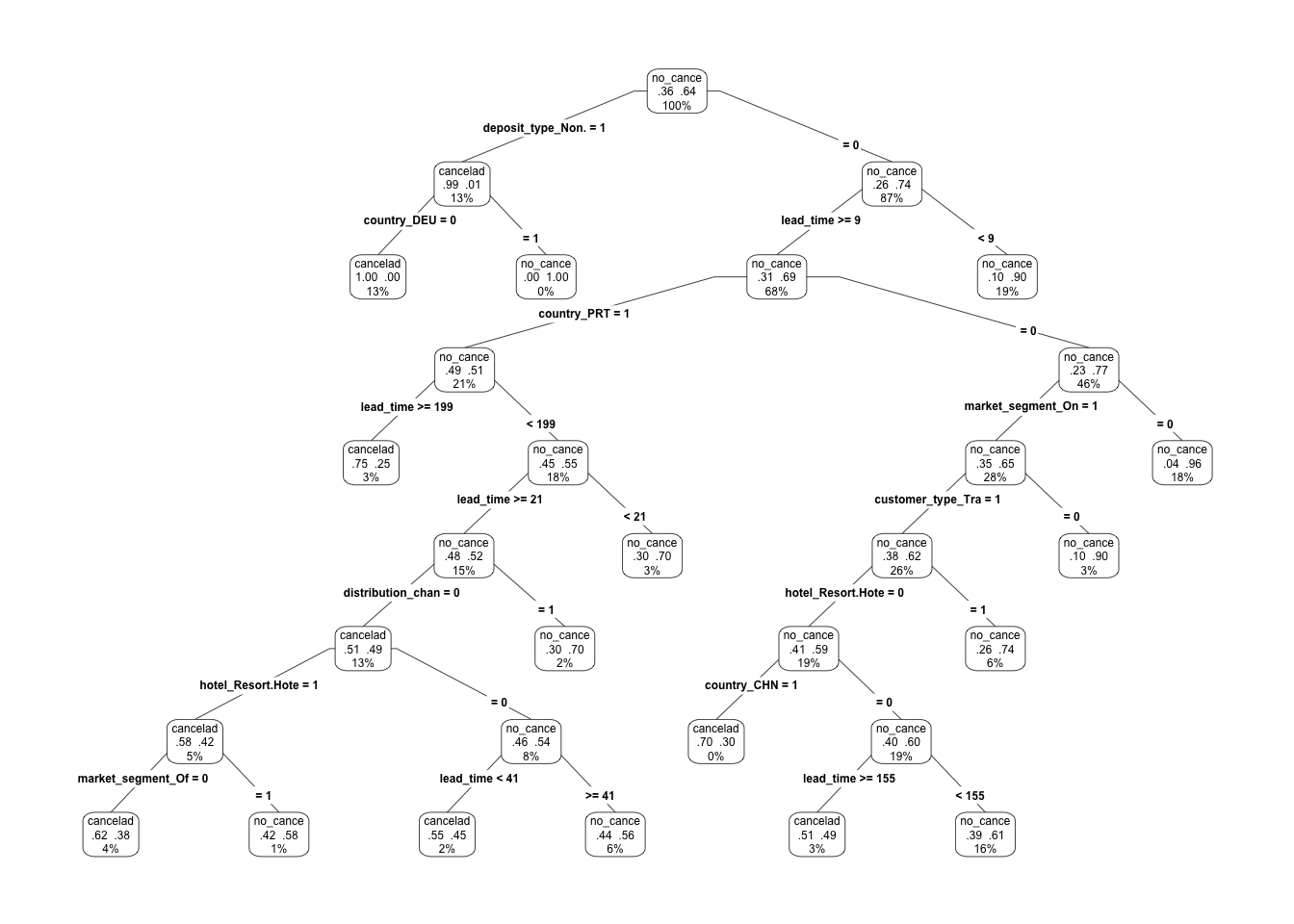
Y vemos cómo dependiendo de las primeras decisiones algunas variables se usan y otras no: este árbol captura interacciones, construidas de manera selectiva, y sólo se utilizan en regiones donde aportan discriminación entre clases.
Un árbol particiona el espacio de entradas en rectángulos paralelos a los ejes, y hace predicciones basadas en un modelo simple dentro de cada una de esas particiones.
Por ejemplo:
knitr::include_graphics('./figuras/arboles_2.png')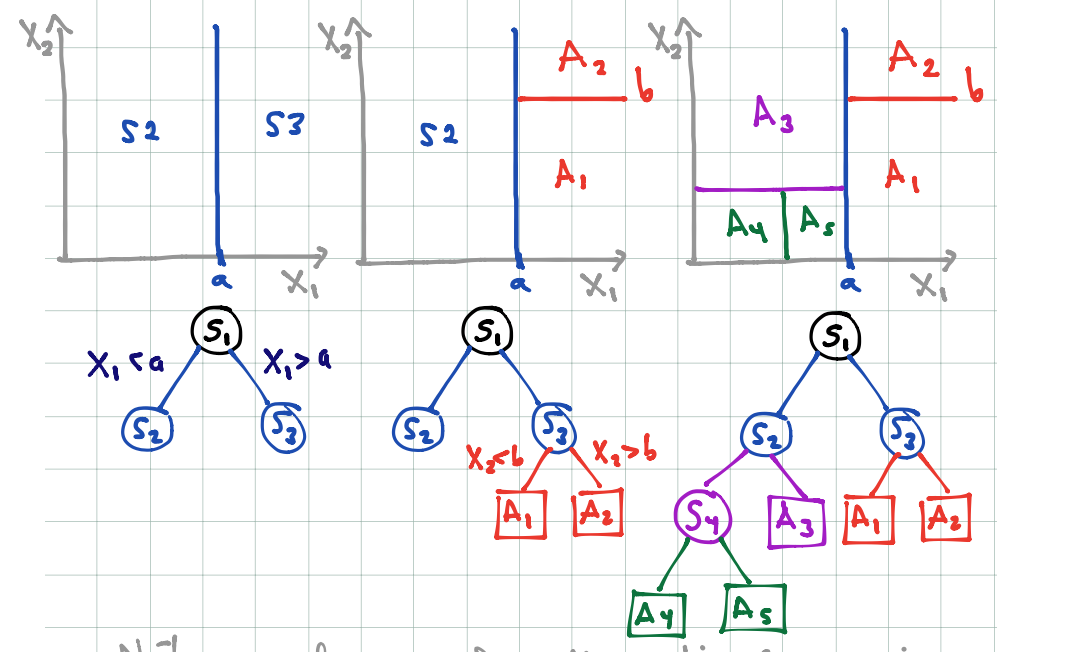
- El proceso de partición binaria recursiva (con una entrada a la vez) puede representarse mediante árboles binarios.
- Los nodos terminales representan a la partición obtenida.
Para definir el proceso de construcción de los árboles, debemos definir:
- ¿Cómo escoger las particiones? Idea: buscar hacer los nodos sucesivamente más puros (que una sola clase domine).
- ¿Cuándo declarar a un nodo como terminal? ¿Cuándo particionar más profundamente? Idea: dependiendo de la aplicación, buscamos hacer árboles chicos, o en otras árboles grandes que después podamos para no sobreajustar.
- ¿Cómo hacer predicciones en nodos terminales? Idea: escoger la clase más común en cada nodo terminal (la de máxima probabilidad).
9.2.1 Tipos de partición
Supongamos que tenemos variables de entrada \((X_1,\ldots, X_p)\). Recursivamente particionamos cada nodo escogiendo entre particiones tales que:
- Dependen de una sola variable de entrada \(X_i\)
- Si \(X_i\) es continua, la partición es de la forma \(\{X_i\leq c\},\{X_i> c\}\), para alguna \(c\) (punto de corte)
- Si \(X_i\) es categórica, la partición es de la forma \(\{X_i\in S\},\{X_i\notin S\}\), para algún subconjunto \(S\) de categorías de \(X_i\).
- En cada nodo candidato, escogemos uno de estos cortes para particionar.
¿Cómo escogemos la partición en cada nodo? En cada nodo, la partición se escoge de una manera miope o local, intentando separar las clases lo mejor que se pueda (sin considerar qué pasa en cortes hechos más adelante). En un nodo dado, escogemos la partición que reduce lo más posible su impureza.
9.2.2 Medidas de impureza
Consideramos un nodo \(t\) de un árbol \(T\), y sean \(p_1(t),\ldots, p_K(t)\) las proporciones de casos de \(t\) que caen en cada categoría.
9.2.2.1 Ejemplo
Graficamos la medida de impureza para dos clases:
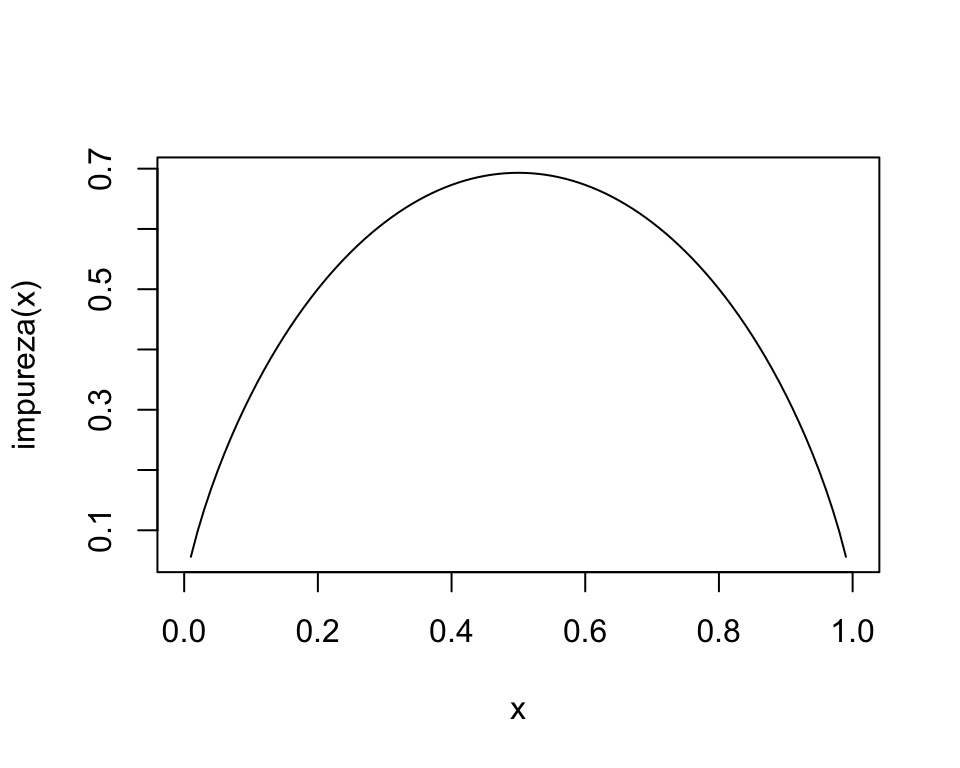
Donde vemos que la máxima impureza se alcanza cuando las proporciones de clase en un nodo son 50-50, y la mínima impureza (máxima pureza) se alcanza cuando en el nodo solo hay casos de una clase. Nótese que esta cantidad es proporcional a la devianza del nodo, donde tenemos porbabilidad constante de clase 1 igual a \(p\).
9.2.3 Reglas de partición y tamaño del árobl
Podemos escribir la regla de partición, que se aplica a cada nodo de un árbol
Ejemplo
Consideremos un nodo \(t\), cuyos casos de entrenamiento son:
## [1] 1.060857Y comparamos con
## [1] 0.7181575Ahora supongamos que tenemos un posible corte, el primero resulta en
n_t <- c(300,10, 140)
n_1 = c(300,0,0)
n_2 = c(0,10,140)
(sum(n_1)/sum(n_t))*impureza(n_1/sum(n_1)) + (sum(n_2)/sum(n_t))*impureza(n_2/sum(n_2))## [1] 0.08164334Un peor corte es:
n_t <- c(300,10, 140)
n_1 = c(200,0,40)
n_2 = c(100,10,100)
(sum(n_1)/sum(n_t))*impureza(n_1/sum(n_1)) + (sum(n_2)/sum(n_t))*impureza(n_2/sum(n_2))## [1] 0.6377053Lo que resta explicar es qué criterio de paro utilizamos para dejar de particionar.
Regla de paro Cuando usemos árboles en ótros métodos, generalmente hay dos opciones:
- Particionar hasta cierta profundidad fija (por ejemplo, máximo 8 nodos terminales). Este enfoque generalmente usa árboles relativamente chicos (se usa en boosting de árboles).
- Dejar de particionar cuando encontramos un número mínimo de casos en un nodo (por ejemplo, 5 o 10 casos). Este enfoque resulta en árboles grandes, probablemente sobreajustados (se usa en bosques aleatorios).
Y cuando utilizamos los árboles por sí solos para hacer predicciones:
- Poda costo complejidad: podemos usar el método CART de Breiman, que consiste en construir un árbol grande y luego podar al tamaño correcto.
Ejemplo
Construímos algunos árboles con los datos de spam:
library(rpart)
library(rpart.plot)
spam_entrena <- read_csv('./datos/spam-entrena.csv') |>
mutate(spam = ifelse(spam == 0, "no_spam", "spam")) |>
mutate(spam = factor(spam))
spam_prueba <- read_csv('./datos/spam-prueba.csv') |>
mutate(spam = ifelse(spam == 0, "no_spam", "spam")) |>
mutate(spam = factor(spam))
head(spam_entrena)## # A tibble: 6 × 59
## ...1 wfmake wfaddress wfall wf3d wfour wfover wfremove wfinternet wforder
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1 0 0.57 0 0 0 0 0 0 0
## 2 2 1.24 0.41 1.24 0 0 0 0 0 0
## 3 3 0 0 0 0 0 0 0 0 0
## 4 4 0 0 0.48 0 0.96 0 0 0 0.48
## 5 5 0.54 0 0.54 0 1.63 0 0 0 0
## 6 6 0 0 0 0 0 0 0 0 0
## # … with 49 more variables: wfmail <dbl>, wfreceive <dbl>, wfwill <dbl>,
## # wfpeople <dbl>, wfreport <dbl>, wfaddresses <dbl>, wffree <dbl>,
## # wfbusiness <dbl>, wfemail <dbl>, wfyou <dbl>, wfcredit <dbl>, wfyour <dbl>,
## # wffont <dbl>, wf000 <dbl>, wfmoney <dbl>, wfhp <dbl>, wfhpl <dbl>,
## # wfgeorge <dbl>, wf650 <dbl>, wflab <dbl>, wflabs <dbl>, wftelnet <dbl>,
## # wf857 <dbl>, wfdata <dbl>, wf415 <dbl>, wf85 <dbl>, wftechnology <dbl>,
## # wf1999 <dbl>, wfparts <dbl>, wfpm <dbl>, wfdirect <dbl>, wfcs <dbl>, …Podemos construir un árbol grande:
spam_arbol <- decision_tree(cost_complexity = 0,
min_n = 1) |>
set_engine("rpart") |>
set_mode("classification") |>
set_args(model = TRUE)
# receta
spam_receta <- recipe(spam ~ ., spam_entrena) |>
step_relevel(spam, ref_level = "no_spam", skip = TRUE)
# flujo
spam_flujo_1 <- workflow() |>
add_recipe(spam_receta) |>
add_model(spam_arbol)
arbol_grande <- fit(spam_flujo_1, spam_entrena)
arbol_grande_1 <- extract_fit_engine(arbol_grande)
prp(arbol_grande_1, type=4, extra=4)## Warning: labs do not fit even at cex 0.15, there may be some overplotting
Podemos examinar la parte de arriba del árbol:
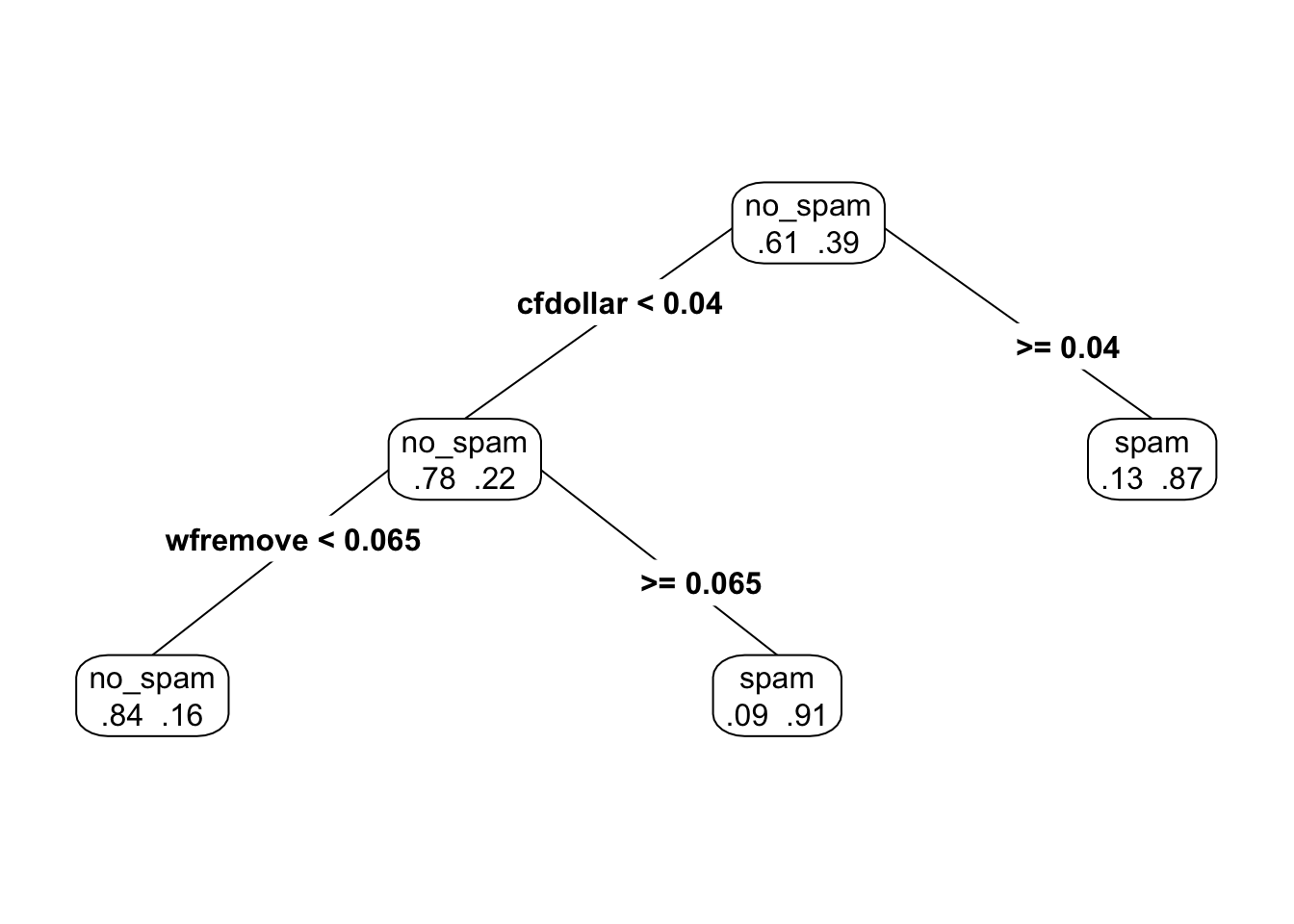
Podemos hacer predicciones con este árbol grande. Por ejemplo, en entrenamiento tenemos las predicciones de clase dan:
metricas_spam <- metric_set(roc_auc, accuracy, sens, spec)
preds_entrena <- predict(arbol_grande, spam_entrena, type = "prob") |>
bind_cols(predict(arbol_grande, spam_entrena)) |>
bind_cols(spam_entrena |> select(spam))
preds_entrena |>
metricas_spam(spam, .pred_no_spam, estimate = .pred_class) |>
mutate(across(is_double, round, 2))## Warning: Predicate functions must be wrapped in `where()`.
##
## # Bad
## data %>% select(is_double)
##
## # Good
## data %>% select(where(is_double))
##
## ℹ Please update your code.
## This message is displayed once per session.## # A tibble: 4 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 accuracy binary 1
## 2 sens binary 1
## 3 spec binary 1
## 4 roc_auc binary 1y en prueba:
preds_prueba <- predict(arbol_grande, spam_prueba, type = "prob") |>
bind_cols(predict(arbol_grande, spam_prueba)) |>
bind_cols(spam_prueba |> select(spam))
preds_prueba |>
metricas_spam(spam, .pred_no_spam, estimate = .pred_class) |>
mutate(across(is_double, round, 2)) ## # A tibble: 4 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 accuracy binary 0.9
## 2 sens binary 0.92
## 3 spec binary 0.88
## 4 roc_auc binary 0.9Y notamos la brecha grande entre prueba y entrenamiento, lo que sugiere sobreajuste. Este árbol es demasiado grande.
9.2.4 Costo - Complejidad (Breiman)
Una manera de escoger árboles del tamaño correcto es utilizando una medida inventada por Breiman para medir la calidad de un árbol. La complejidad de un árbol \(T\) está dada por (para \(\alpha\) fija):
\[C_\alpha (T) = \overline{err}(T) + \alpha \vert T\vert\] donde
- \(\overline{err}(T)\) es el error de clasificación de \(T\)
- \(\vert T\vert\) es el número de nodos terminales del árbol
- \(\alpha>0\) es un parámetro de penalización del tamaño del árbol.
Esta medida de complejidad incluye qué tan bien clasifica el árbol en la muestra de entrenamiento, pero penaliza por el tamaño del árbol.
Para escoger el tamaño del árbol correcto, definimos \(T_\alpha \subset T\) como el subárbol de \(T\) que minimiza la medida \(C_\alpha (T_\alpha)\).
Para entender esta decisión, obsérvese que:
- Un subárbol grande de \(T\) tiene menor valor de \(\overline{err}(T)\) (pues usa más cortes)
- Pero un subárbol grande de \(T\) tiene más penalización por complejidad \(\alpha\vert T\vert\).
De modo que para \(\alpha\) fija, el árbol \(T_\alpha\) hace un balance entre error de entrenamiento y penalización por complejidad.
9.2.4.1 Ejemplo
Podemos ver subárboles más chicos creados durante el procedimiento de división de nodos (prp está el paquete rpart.plot). En este caso pondemos \(\alpha = 0.2\) (cp = \(\alpha\) = complexity parameter):
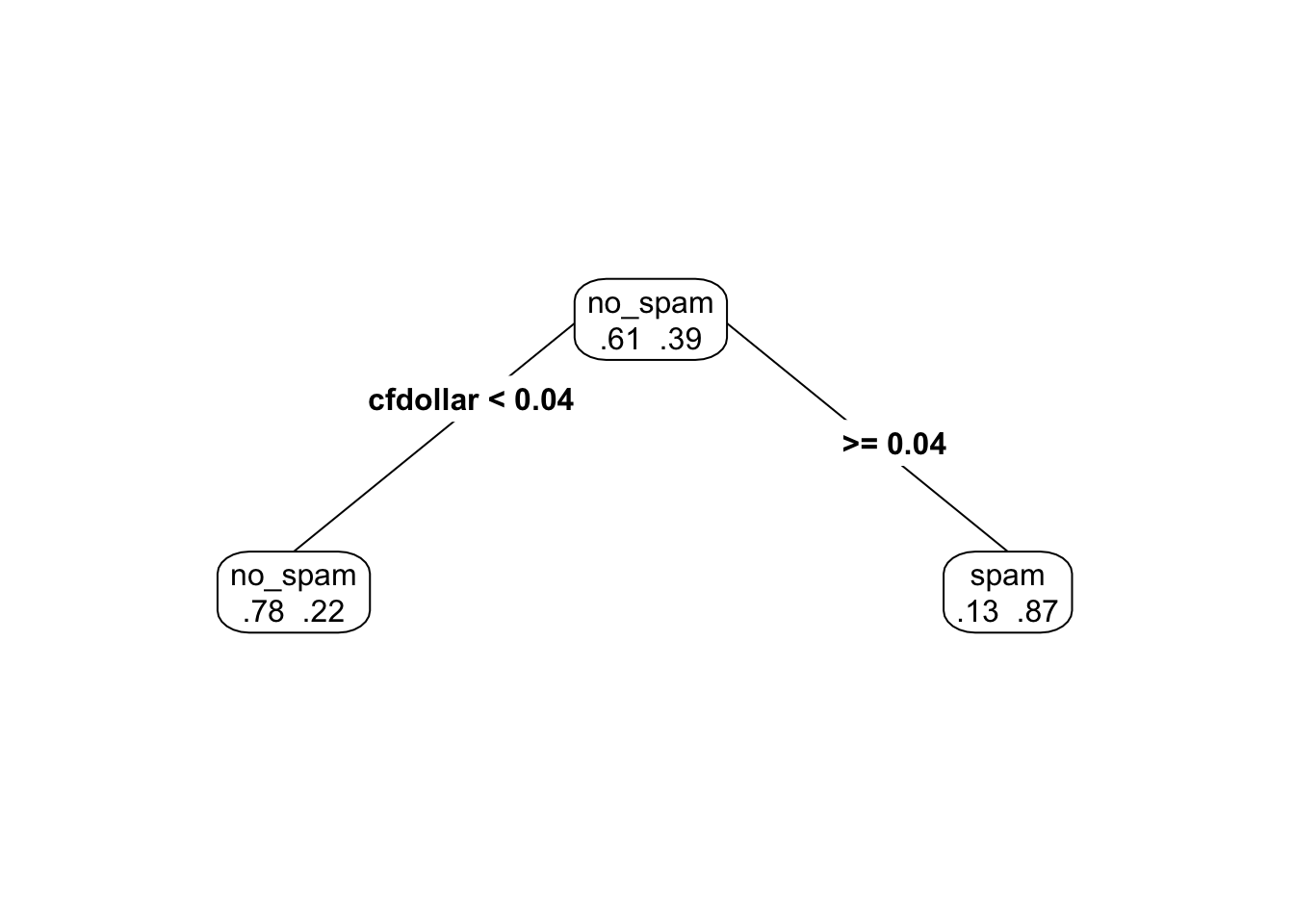
Si disminuimos el coeficiente \(\alpha\).
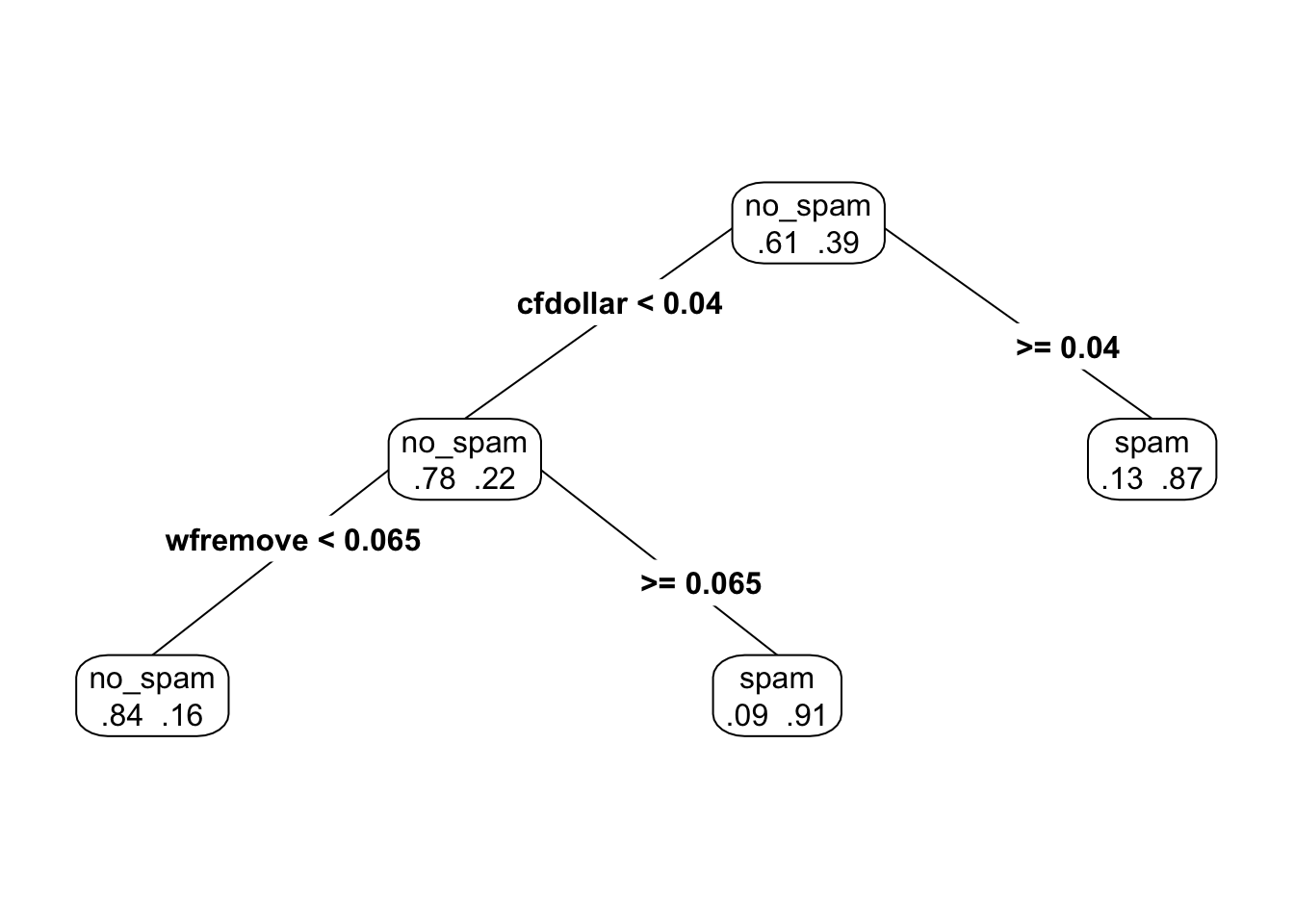
y vemos que en efecto el árbol \(T_{0.07}\) contiene al árbol \(T_{0.2}\), y ambos son subárboles del árbol gigante que construimos al principio.
Nota: Esto es un teorema que hace falta demostrar: el resultado principal es que conforme aumentamos \(\alpha\), vamos eliminiando ramas del árbol, de manera que los árboles más chicos siempre sin subárboles de los más grandes.
source('./R/fancyRpartPlot.R')
fancyRpartPlot(arbol_chico_1, sub='')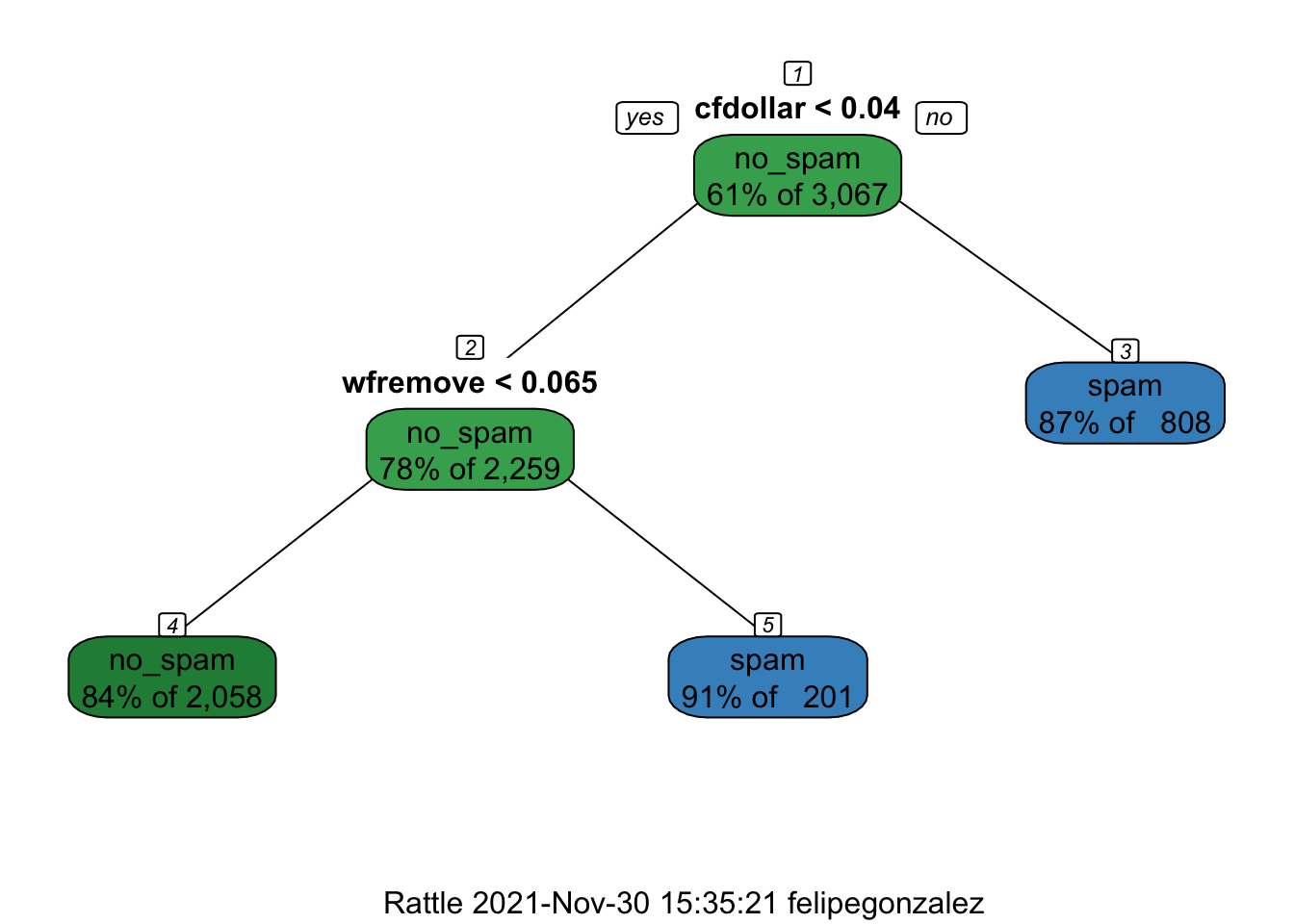
Nota: Enfoques de predicción basados en un solo árbol para clasificación y regresión son típicamente superados en predicción por otros métodos. ¿Cuál crees que sea la razón? ¿Es un problema de varianza o sesgo?
9.2.5 Predicciones con CART
En el método de poda usual y selección de complejidad seleccionamos la complejidad que minimiza el error de clasificación.
# esta es una manera de que la validación cruzada
# corra en paralelo.
# install.packages("doParallel")
# install.packages("doFuture")
library(doParallel)
library(doFuture)
registerDoFuture()
cl <- makeCluster(4)
plan(cluster, workers = cl)
set.seed(993) # para hacer reproducible la validación cruzada
cortes_vc <- vfold_cv(spam_entrena, v = 10)
# afinamos dos parámetros
spam_arbol <- decision_tree(cost_complexity = tune(),
min_n = tune()) |>
set_engine("rpart") |>
set_mode("classification")
spam_receta <- recipe(spam ~ ., spam_entrena)
spam_flujo <- workflow() |>
add_recipe(spam_receta) |>
add_model(spam_arbol)
# validación cruzada
valores_grid <- expand_grid(cost_complexity = c(exp(seq(-8, -4, 0.25))),
min_n = c(5, 10, 20, 40))
evaluacion_vc <- tune_grid(spam_flujo,
resamples = cortes_vc,
grid = valores_grid)
metricas_vc <- collect_metrics(evaluacion_vc)
metricas_vc## # A tibble: 136 × 8
## cost_complexity min_n .metric .estimator mean n std_err .config
## <dbl> <dbl> <chr> <chr> <dbl> <int> <dbl> <chr>
## 1 0.000335 5 accuracy binary 0.919 10 0.00538 Preprocessor1_…
## 2 0.000335 5 roc_auc binary 0.922 10 0.00801 Preprocessor1_…
## 3 0.000335 10 accuracy binary 0.918 10 0.00599 Preprocessor1_…
## 4 0.000335 10 roc_auc binary 0.941 10 0.00626 Preprocessor1_…
## 5 0.000335 20 accuracy binary 0.910 10 0.00537 Preprocessor1_…
## 6 0.000335 20 roc_auc binary 0.946 10 0.00433 Preprocessor1_…
## 7 0.000335 40 accuracy binary 0.895 10 0.00514 Preprocessor1_…
## 8 0.000335 40 roc_auc binary 0.936 10 0.00572 Preprocessor1_…
## 9 0.000431 5 accuracy binary 0.920 10 0.00546 Preprocessor1_…
## 10 0.000431 5 roc_auc binary 0.923 10 0.00807 Preprocessor1_…
## # … with 126 more rowsY vemos los resultados:
ggplot(metricas_vc |> filter(.metric =="roc_auc"),
aes(x = cost_complexity, y = mean,
ymin = mean - std_err, ymax = mean + std_err, group = factor(min_n),
colour = factor(min_n))) +
geom_linerange() +
geom_line() +
geom_point() +
scale_x_log10() +
ylab("AUC estimado vc-10")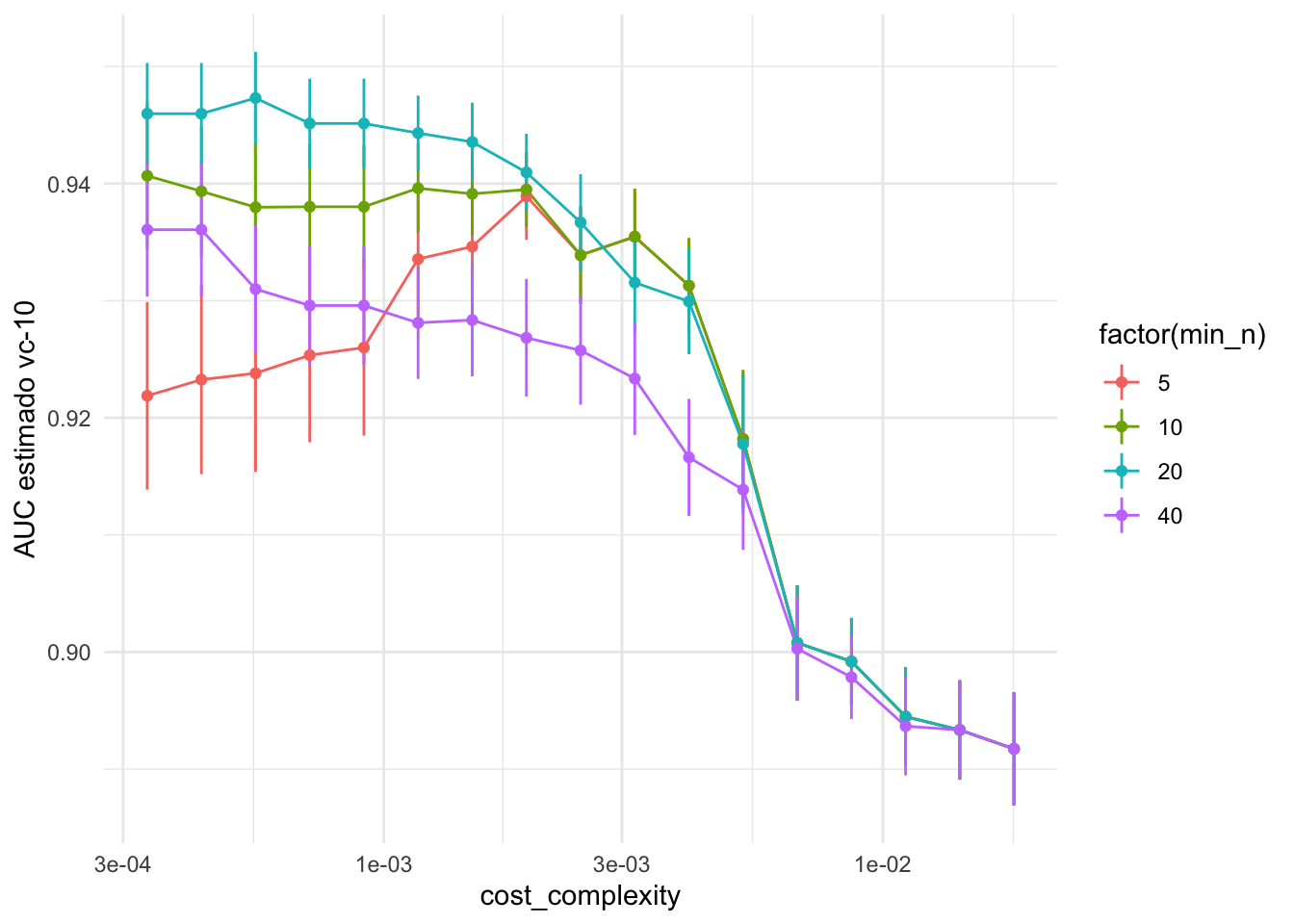
Y usamos la regla de mínimo error o a una desviación estándar del error mínimo:
mejor_arbol <- select_by_one_std_err(evaluacion_vc,
metric = "roc_auc", desc(cost_complexity))
mejor_arbol## # A tibble: 1 × 10
## cost_complexity min_n .metric .estimator mean n std_err .config .best
## <dbl> <dbl> <chr> <chr> <dbl> <int> <dbl> <chr> <dbl>
## 1 0.00150 20 roc_auc binary 0.944 10 0.00335 Preprocess… 0.947
## # … with 1 more variable: .bound <dbl>Y ajustamos el modelo final y lo evaluamos:
arbol_podado_vc <- finalize_workflow(spam_flujo, mejor_arbol) |>
fit(spam_entrena)
predict(arbol_podado_vc, spam_prueba, type = "prob") |>
bind_cols(predict(arbol_podado_vc, spam_prueba)) |>
bind_cols(spam_prueba |> select(spam)) |>
metricas_spam(spam, .pred_no_spam, estimate = .pred_class) |>
mutate(across(is_double, round, 2))## # A tibble: 4 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 accuracy binary 0.91
## 2 sens binary 0.94
## 3 spec binary 0.87
## 4 roc_auc binary 0.939.2.6 Árboles para regresión
Para problemas de regresión, el criterio de pureza y la predicción en cada nodo terminal es diferente:
- En los nodos terminales usamos el promedio los casos de entrenamiento que caen en tal nodo (en lugar de la clase más común)
- La impureza de define como varianza: si \(t\) es un nodo, su impureza está dada por \(\frac{1}{n(t)}\sum (y - m)^2\), donde la suma es sobre los casos que están en el nodo y \(m\) es la media de las \(y\)’s del nodo.
9.2.7 Variabilidad en el proceso de construcción
Existe variabilidad considerable en el proceso de división, lo cual es una debilidad de los árboles. Por ejemplo:
# muestra bootstrap
set.seed(91923)
muestra_1 <- sample_frac(spam_entrena, 1 , replace = TRUE)
spam_1 <-rpart(spam ~ ., data = muestra_1, method = "class")
arbol_podado <- prune(spam_1, cp=0.03)
prp(arbol_podado, type = 4, extra = 4)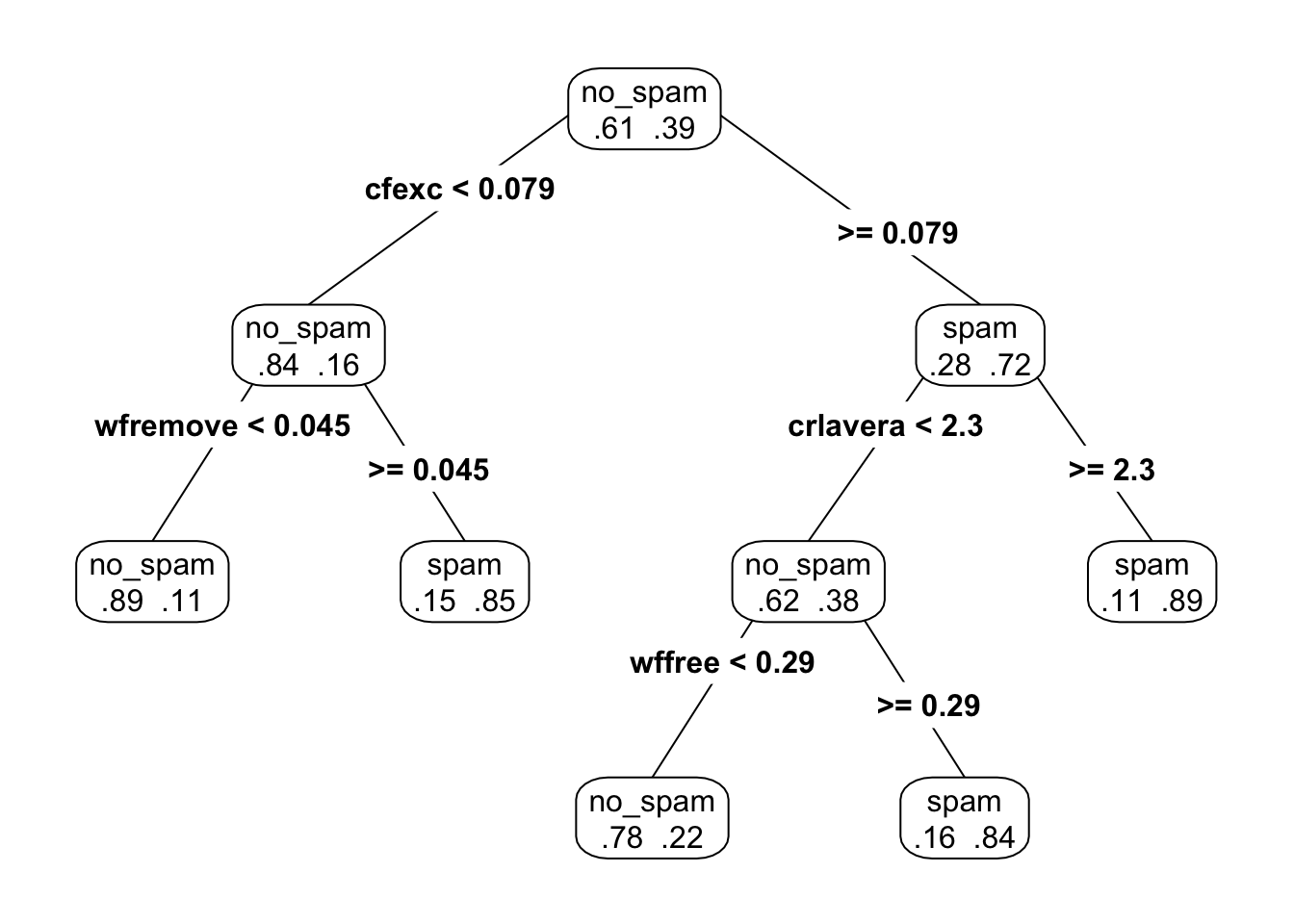
# muestra bootstrap
muestra_1 <- sample_frac(spam_entrena, 1 , replace = TRUE)
spam_1 <-rpart(spam ~ ., data = muestra_1, method = "class")
arbol_podado <- prune(spam_1, cp=0.03)
prp(arbol_podado, type = 4, extra = 4)
Pequeñas diferencias en la muestra de entrenamiento produce distintas selecciones de variables y puntos de corte, y estructuras de árboles muchas veces distintas. Esto introduce varianza considerable en las predicciones.
9.2.8 Relaciones lineales
Los árboles pueden requerir ser muy grandes para estimar apropiadamente relaciones lineales.
x <- runif(200, 0, 1)
y <- 2*x + rnorm(200, 0, 0.1)
arbol <- rpart(y ~ x, data = tibble(x = x, y = y), method = 'anova')
x_pred <- seq(0, 1, 0.05)
y_pred <- predict(arbol, newdata = tibble(x = x_pred))
y_verdadera <- 2 * x_pred
dat <- tibble(x_pred = x_pred, y_pred = y_pred, y_verdadera = y_verdadera) |>
pivot_longer(cols = y_pred:y_verdadera, "y")
ggplot(dat, aes(x = x_pred, y = value, colour = y)) + geom_line() 
9.2.9 Ventajas y desventajas de árboles
Ventajas:
- Árboles chicos son relativamente fáciles de explicar
- Capturan interacciones entre las variables de entrada
- Son robustos en el sentido de que
- valores numéricos atípicos no hacen fallar al método
- no es necesario transformar (monótonamente) variables de entrada
- hay formas fáciles de lidiar con datos faltantes (cortes sucedáneos)
- Se ajustan rápidamente y son relativamente fáciles de interpretar (por ejemplo, son útiles para clasificar en campo)
- Árboles grandes generalmente no sufren de sesgo.
Desventajas:
- Tienen dificultades en capturar estructuras lineales.
- En la interpretación, tienen la dificultad de que muchas veces algunas variables de entrada “enmascaran” a otras. Que una variable de entrada no esté en el árbol no quiere decir que no sea “importante” para predecir (regresión ridge lidia mejor con esto).
- Son inestables (varianza alta) por construcción: es local/miope, basada en cortes duros si/no. Esto produce desempeño predictivo relativamente malo. (p ej: una pequeña diferencia en cortes iniciales puede resultar en estructuras de árbol totalmente distintas).
- Adicionalmente, no son apropiados cuando hay variables categóricas con muchas niveles: en estos casos, el árbol sobreajusta desde los primeros cortes, y las predicciones son malas.
9.3 Bagging de árboles
Bosques aleatorios es un método de predicción que utiliza familias de árboles para hacer predicciones.
Los árboles grandes tienen la ventaja de tener sesgo bajo, pero sufren de varianza alta. Podemos explotar el sesgo bajo si logramos controlar la varianza. Una idea primera para lograr esto es es hacer bagging de árboles:
- Perturbar la muestra de entrenamiento de distintas maneras y producir árboles distintos (grandes). La perturbación más usada es tomar muestras bootstrap de los datos y ajustar un árbol a cada muestra bootstrap
- Promediar el resultado de todos estos árboles para hacer predicciones. El proceso de promediar reduce la varianza, sin tener pérdidas en sesgo.
La idea básica de bagging (bootstrap aggregation) es la siguiente:
Consideramos el proceso \({\mathcal L} \to T_{\mathcal L}\), que representa el proceso de ajuste de un árbol \(T_{\mathcal L}\) a partir de la muestra de entrenamiento \({\mathcal L}\). Si pudiéramos obtener distintas muestras de entrenamiento \[{\mathcal L}_1, {\mathcal L}_2, \ldots, {\mathcal L}_B,\] y supongamos que construimos los árboles (que suponemos de regresión) \[T_1, T_2, \ldots, T_B,\] Podríamos mejorar nuestras predicciones construyendo el árbol promedio \[T(x) = \frac{1}{B}\sum_{i=b}^B T_b (x)\] ¿Por qué es mejor este árbol promedio que cualquiera de sus componentes? Veamos primero el sesgo. El valor esperado del árbol promedio es \[E[T(x)] = \frac{1}{B}\sum_{i=b}^B E[T_b (x)]\] y como cada \(T_b(x)\) se construye de la misma manera a partir de \({\mathcal L}_b\), y todas las muestras \({\mathcal L}_b\) se extraen de la misma forma, todos los términos de la suma de la derecha son iguales: \[E[T(x)] = E[T_1 (x)],\] lo que implica que el sesgo del promedio es igual al sesgo de un solo árbol (que es bajo, pues suponemos que los árboles son grandes).
Ahora veamos la varianza. Como las muestras \({\mathcal L}_b\) se extraen de manera independiente, entonces
\[Var[T(x)] = Var\left( \frac{1}{B}\sum_{i=b}^B T_b (x)\right) = \frac{1}{B^2}\sum_{i=b}^B Var[T_b (x)],\] pues los distintos \(T_b(x)\) no están correlacionados (en ese caso, varianza de la suma es la suma de las varianzas), y las constantes salen de la varianza al cuadrado. Por las mismas razones que arriba, todos los términos de la derecha son iguales, y \[Var[T(x)] = \frac{1}{B}\ Var[T_1 (x)]\] de modo que la varianza del árbol promedio es mucho más chica que la varianza de un árbol dado (si \(B\) es grande).
Sin embargo, no podemos tomar muestras de entrenamiento repetidamente para ajustar estos árboles. ¿Cómo podemos simular extraer distintas muestras de entrenamiento?
Sabemos que si tenemos una muestra de entrenamiento fija \({\mathcal L}\), podemos evaluar la variación de esta muestra tomando muestras bootstrap de \({\mathcal L}\), que denotamos por
\[{\mathcal L}_1^*, {\mathcal L}_2^*, \ldots, {\mathcal L}_B^*,\]
Recordatorio: una muestra bootstrap de \(\mathcal L\) es una muestra con con reemplazo de \({\mathcal L}\) del mismo tamaño que \({\mathcal L}\).Entonces la idea es que construimos los árboles (que suponemos de regresión) \[T_1^*, T_2^*, \ldots, T_B^*,\] podríamos mejorar nuestras predicciones construyendo el árbol promedio \[T^*(x) = \frac{1}{B}\sum_{i=b}^B T_b^* (x)\] para suavizar la variación de cada árbol individual.
El argumento del sesgo aplica en este caso, pero el de la varianza no exactamente, pues las muestras bootstrap no son independientes (están correlacionadas a través de la muestra de entrenamiento de donde se obtuvieron),a pesar de que las muestras bootstrap se extraen de manera independiente de \({\mathcal L}\). De esta forma, no esperamos una reducción de varianza tan grande como en el caso de muestras independientes.
Bagging Sea \({\mathcal L} =\{(x^{(i)}, y^{(i)})\}_{i=1}^n\) una muestra de entrenamiento, y sean \[{\mathcal L}_1^*, {\mathcal L}_2^*, \ldots, {\mathcal L}_B^*,\] muestras bootstrap de \({\mathcal L}\) (muestreamos con reemplazo los pares \((x^{(i)}, y^{(i)})\), para obtener una muestra de tamaño \(n\)).
- Para cada muestra bootstrap construimos un árbol \[{\mathcal L}_b^* \to T_b^*\].
- (Regresión) Promediamos árboles para reducir varianza \[T^*(x) = \frac{1}{B}\sum_{i=b}^B T_b^*(x)\]
- (Clasificación) Tomamos votos sobre todos los árboles: \[T^*(x) = argmax_g \{ \# \{i|T_b^*(x)=g\}\}.\] Podemos también calcular probabilidades promedio sobre todos los árboles.
Nota: No hay garantía de bagging reduzca el error de entrenamiento, especialmente si los árboles base son muy malos clasificadores ¿Puedes pensar en un ejemplo donde empeora?
9.3.1 Ejemplo
Probemos con el ejemplo de spam. Construimos árboles con muestras bootstrap de los datos originales de entrenamiento:
muestra_bootstrap <- function(df){
df |> sample_n(nrow(df), replace = TRUE)
}
modelo_spam <- decision_tree(cost_complexity = 0, min_n = 5) |>
set_engine("rpart") |>
set_mode("classification") |>
set_args(model = TRUE)
# crear 30 árboles con muestras bootstrap
arboles_bagged <- map(1:30, function(i){
muestra <- muestra_bootstrap(spam_entrena)
arbol <- modelo_spam |> fit(spam ~ ., data = muestra)
arbol$fit
})Examinemos la parte de arriba de algunos de estos árboles:
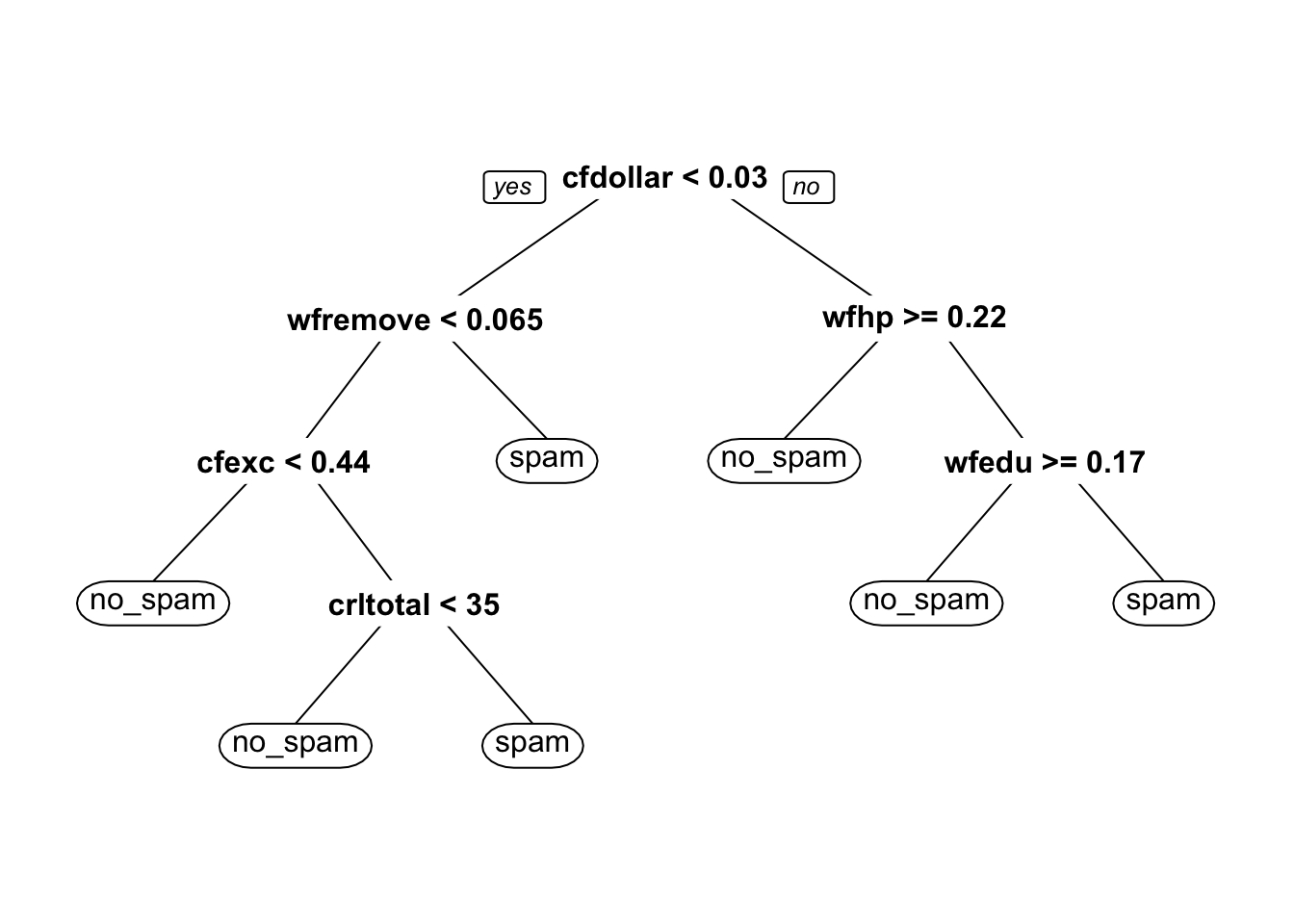
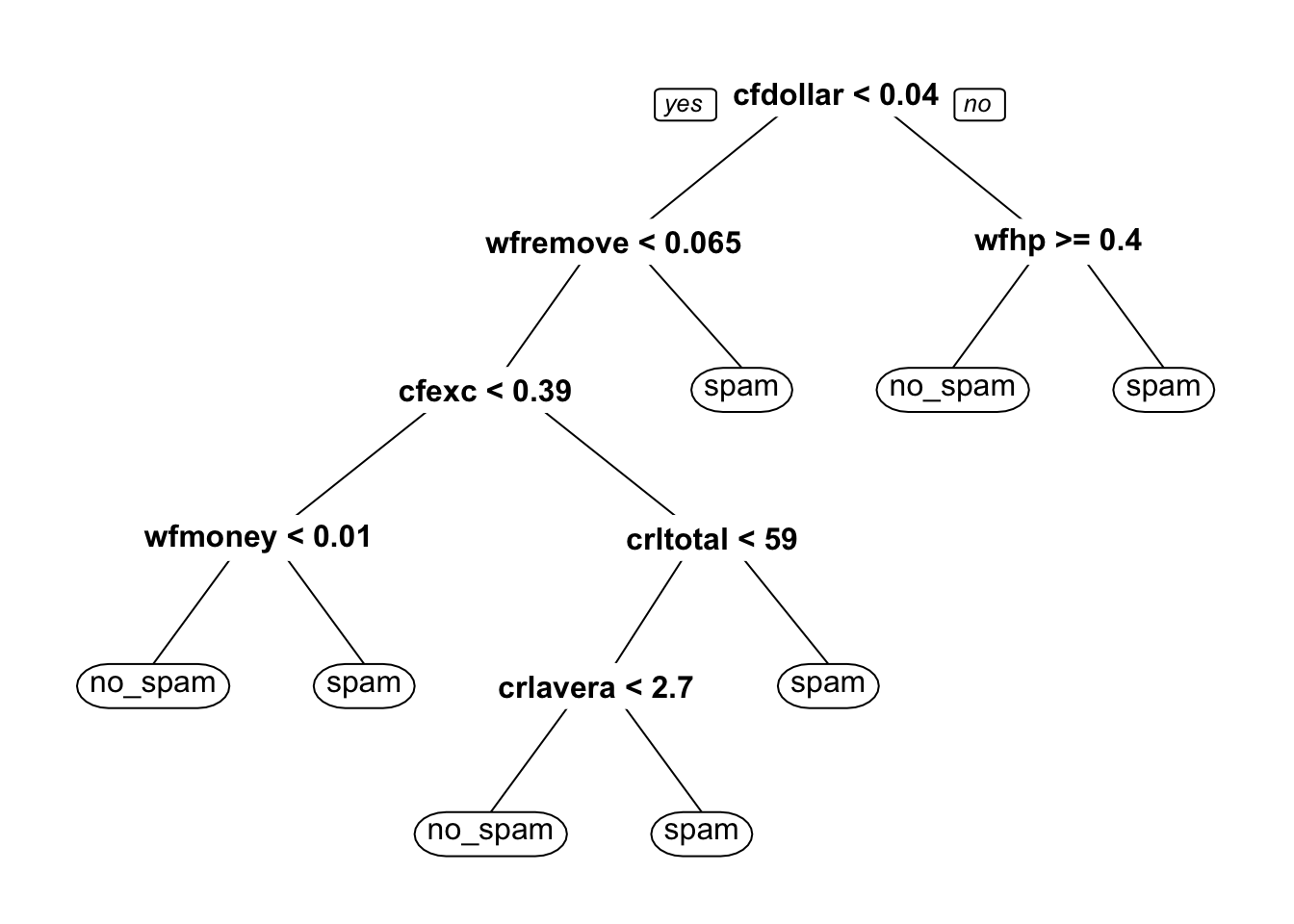
Ahora probemos hacer predicciones con los 30 árboles. Haremos el bagging manualmente:
preds_clase_1 <- map(arboles_bagged, function(arbol){
preds <- predict(arbol, spam_prueba, type = "prob")[, 2]
})
preds <- preds_clase_1 |> as_tibble(.name_repair = "unique") |>
mutate(id = row_number())
dim(preds)## [1] 1534 31
prob_bagging <- preds |>
pivot_longer(cols = -id, names_to = "arbol", values_to = "prob") |>
group_by(id) |>
summarise(prob = mean(prob)) |>
bind_cols(spam_prueba |> select(spam))
roc_auc(prob_bagging, truth = spam, estimate = prob, event_level = "second")## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 roc_auc binary 0.975
table(prob_bagging$prob > 0.5, prob_bagging$spam) |>
prop.table(2) |> round(2)##
## no_spam spam
## FALSE 0.96 0.10
## TRUE 0.04 0.90Y vemos que tenemos una mejora inmediata con respecto un sólo árbol grande (tanto un árbol grande como uno podado con costo-complejidad). El único costo es el cómputo adicional para procesar las muestras bootstrap.
Podemos hacerlo automáticamente de las siguiente forma:
library(baguette)
set.seed(123)
arboles_bag <- bag_tree(cost_complexity = 0, min_n = 5) |>
set_engine("rpart", times = 30) |>
set_mode("classification") |>
fit(spam ~ ., spam_entrena)
predict(arboles_bag, spam_prueba) |>
bind_cols(predict(arboles_bag, spam_prueba, type = "prob")) |>
bind_cols(spam_prueba |> select(spam)) |>
metricas_spam(spam, .pred_no_spam, estimate = .pred_class) |>
mutate(across(is_double, round, 2))## # A tibble: 4 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 accuracy binary 0.94
## 2 sens binary 0.96
## 3 spec binary 0.9
## 4 roc_auc binary 0.98- ¿Cuántas muestras bootstrap? Bagging generalmente funciona mejor cuando tomamos tantas muestras como sea posible - aunque también es un parámetro que se puede afinar.
- Bagging por sí solo se usa rara vez. El método más poderoso es bosques aleatorios, donde el proceso básico es bagging de árboles, pero añadimos ruido adicional en la construcción de árboles.
9.3.2 Mejorando bagging
El factor que limita la mejora de desempeño de bagging es que los árboles están correlacionados a través de la muestra de entrenamiento. Como vimos, si los árboles fueran independientes, entonces mejoramos por un factor de \(B\) (número de muestras independientes). Veamos un argumento para entender cómo esa correlación limita las mejoras:
Quiséramos calcular (para una \(x\) fija)
\[Var(T(x)) = Var\left(\frac{1}{B}\sum_{i=1}^B T^*_i\right)\]
donde cada \(T^*_i\) se construye a partir de una muestra bootstrap de \({\mathcal L}\). Nótese que esta varianza es sobre la muestra de entrenamiento \({\mathcal L}\). Usando la fórmula de la varianza para sumas generales: \[\begin{equation} Var(T(x)) = Var\left(\frac{1}{B}\sum_{i=1}^B T^*_i\right) = \sum_{i=1}^B \frac{1}{B^2} Var(T^*_i(x)) + \frac{2}{B^2}\sum_{i < j} Cov(T_i^* (x), T_j^* (x)) \tag{9.1} \end{equation}\]
Ponemos ahora
\[\sigma^2(x) = Var(T_i^* (x))\] que son todas iguales porque los árboles bootstrap se extraen de la misma manera (\({\mathcal L}\to {\mathcal L}^*\to T^*\)).
Escribimos ahora \[\rho(x) = corr(T_i^* (x), T_j^* (x))\] que es una correlación sobre \({\mathcal L}\) (asegúrate que entiendes este término). Todas estas correlaciones son iguales pues cada par de árboles se construye de la misma forma.
Así que la fórmula (9.1) queda
\[\begin{equation} Var(T(x)) = \frac{1}{B} \sigma^2(x) + \frac{B-1}{B} \rho(x)\sigma^2(x) = \sigma^2(x)\left(\frac{1}{B} + \left(1-\frac{1}{B}\right )\rho(x) \right) \tag{9.2} \end{equation}\]
En el límite (cuando B es muy grande, es decir, promediamos muchos árboles):
\[\begin{equation} Var(T(x)) = Var\left(\frac{1}{B}\sum_{i=1}^B T^*_i\right) \approx \sigma^2(x)\rho(x) \tag{9.3} \end{equation}\]
Si \(\rho(x)=0\) (árboles no correlacionados), la varianza del ensemble es la fracción \(1/B\) de la varianza de un solo árbol, y obtenemos una mejora considerable en varianza. En el otro extremo, si la correlación es alta \(\rho(x)\approx 1\), entonces no obtenemos ganancias por promediar árboles y la varianza del ensamble es similar a la de un solo árbol.
- Cuando hacemos bagging de árboles, la limitación de mejora cuando promediamos muchos árboles está dada por la correlación entre ellos: cuanto más grande es la correlación, menor beneficio en reducción de varianza obtenemos.
- Si alteramos el proceso para producir árboles menos correlacionados (menor \(\rho(x)\)), podemos mejorar el desempeño de bagging. Sin embargo, estas alteraciones generalmente están acompañadas de incrementos en la varianza (\(\sigma^x(x)\)).
9.4 Bosques aleatorios
Los bosques aleatorios son una versión de árboles de bagging decorrelacionados. Esto se logra introduciendo variabilidad en la construcción de los árboles (esto es paradójico - pero la explicación está arriba: aunque la varianza empeora (de cada árbol), la decorrelación de árboles puede valer la pena).
El proceso de decorrelación de bosques aleatorios consiste en que cada vez que tengamos que hacer un corte en un árbol de bagging, escoger al azar un número de variables y usar estas para buscar la mejor variable y el mejor punto de corte, como hicimos en la construcción de árboles.
Bosques aleatorios Sea \(m\) fija. Sea \({\mathcal L} =\{(x^{(i)}, y^{(i)})\}_{i=1}^n\) una muestra de entrenamiento, y sean \[{\mathcal L}_1^*, {\mathcal L}_2^*, \ldots, {\mathcal L}_B^*,\] muestras bootstrap de \({\mathcal L}\) (muestreamos con reemplazo los pares \((x^{(i)}, y^{(i)})\), para obtener una muestra de tamaño \(n\)).
- Para cada muestra bootstrap construimos un árbol \[{\mathcal L}_b^* \to T_b^*\] de la siguiente forma:
- En cada nodo candidato a particionar, escogemos al azar \(m\) variables de las disponibles
- Buscamos la mejor variable y punto de corte (como en un árbol normal) pero solo entre las variables que seleccionamos al azar.
- Seguimos hasta construir un árbol grande.
- (Regresión) Promediamos árboles para reducir varianza \[T^*(x) = \frac{1}{B}\sum_{i=b}^B T_b^*(x)\]
- (Clasificación) Tomamos votos sobre todos los árboles: \[T^*(x) = argmax_g \{ \# \{i|T_b^*(x)=g\}\}.\] Podemos también calcular probabilidades promediando sobre todos los árboles las proporciones de clase de cada árbol.
Bosques aleatorios muchas veces reduce el error de predicción gracias a una reducción a veces considerable de varianza. El objetivo final es reducir la varianza alta que producen árboles normales debido a la forma tan agresiva de construir sus cortes.
Observaciones:
- El número de variables \(m\) que se seleccionan en cada nodo es un parámetro que hay que escoger (usando validación, validación cruzada).
- Ojo: no se selecciona un conjunto de \(m\) variables para cada árbol. En la construcción de cada árbol, en cada nodo se seleccionan \(m\) variables como candidatas para cortes.
- Como inducimos aleatoriedad en la construcción de árboles, este proceso reduce la correlación entre árboles del bosque, aunque también incrementa su varianza. Los bosques aleatorios funcionan bien cuando la mejora en correlación es más grande que la pérdida en varianza.
- Reducir \(m\), a grandes rasgos:
- Aumenta el sesgo del bosque (pues es más restringido el proceso de construcción)
- Disminuye la correlación entre árboles y aumenta la varianza de cada árbol
- Incrementar \(m\)
- Disminuye el sesgo del bosque (menos restricción)
- Aumenta la correlacción entre árobles y disminuye la varianza de cada árbol
- Cuando usamos bosques aleatorios para estimar probabilidades de clase, como siempre, es necesario checar la calibración de esas probabilidades (ver sección de regresión logística).
Ejemplo
Regresamos a nuestro ejemplo de spam. Intentemos con 500 árboles, y 6 variables (de 58 variables) para escoger como candidatos en cada corte:
spam_bosque <- rand_forest(mtry = 6, trees = 1000) |>
set_engine("ranger", importance = "permutation") |>
set_mode("classification")
# flujo
spam_flujo_2 <- workflow() |>
add_recipe(spam_receta) |>
add_model(spam_bosque)
flujo_ajustado <- fit(spam_flujo_2, spam_entrena)
bosque <- extract_fit_parsnip(flujo_ajustado)Evaluamos desempeño, donde vemos que obtenemos una mejora inmediata con respecto a bagging:
predict(flujo_ajustado , spam_prueba, type = "prob") |>
bind_cols(predict(flujo_ajustado, spam_prueba)) |>
bind_cols(spam_prueba |> select(spam)) |>
metricas_spam(spam, .pred_no_spam, estimate = .pred_class) |>
mutate(across(is_double, round, 2))## # A tibble: 4 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 accuracy binary 0.95
## 2 sens binary 0.97
## 3 spec binary 0.91
## 4 roc_auc binary 0.98Comparemos las curvas ROC para:
- árbol grande sin podar
- árbol podado con costo-complejidad
- bagging de árboles
- bosque aleatorio
Las curvas de precision-recall:
modelos <- list(arbol_grande = arbol_grande,
podado = arbol_podado_vc,
bagging = arboles_bag,
bosque = bosque)
prec_tbl <- map(names(modelos), function(mod_nombre){
predict(modelos[[mod_nombre]], spam_prueba, type = "prob") |>
bind_cols(spam_prueba |> select(spam)) |>
pr_curve(spam, .pred_no_spam) |>
mutate(modelo = mod_nombre)
}) |>
bind_rows()
ggplot(prec_tbl,
aes(x = recall, y = precision, colour = modelo)) +
geom_path() + geom_point(size = 1) 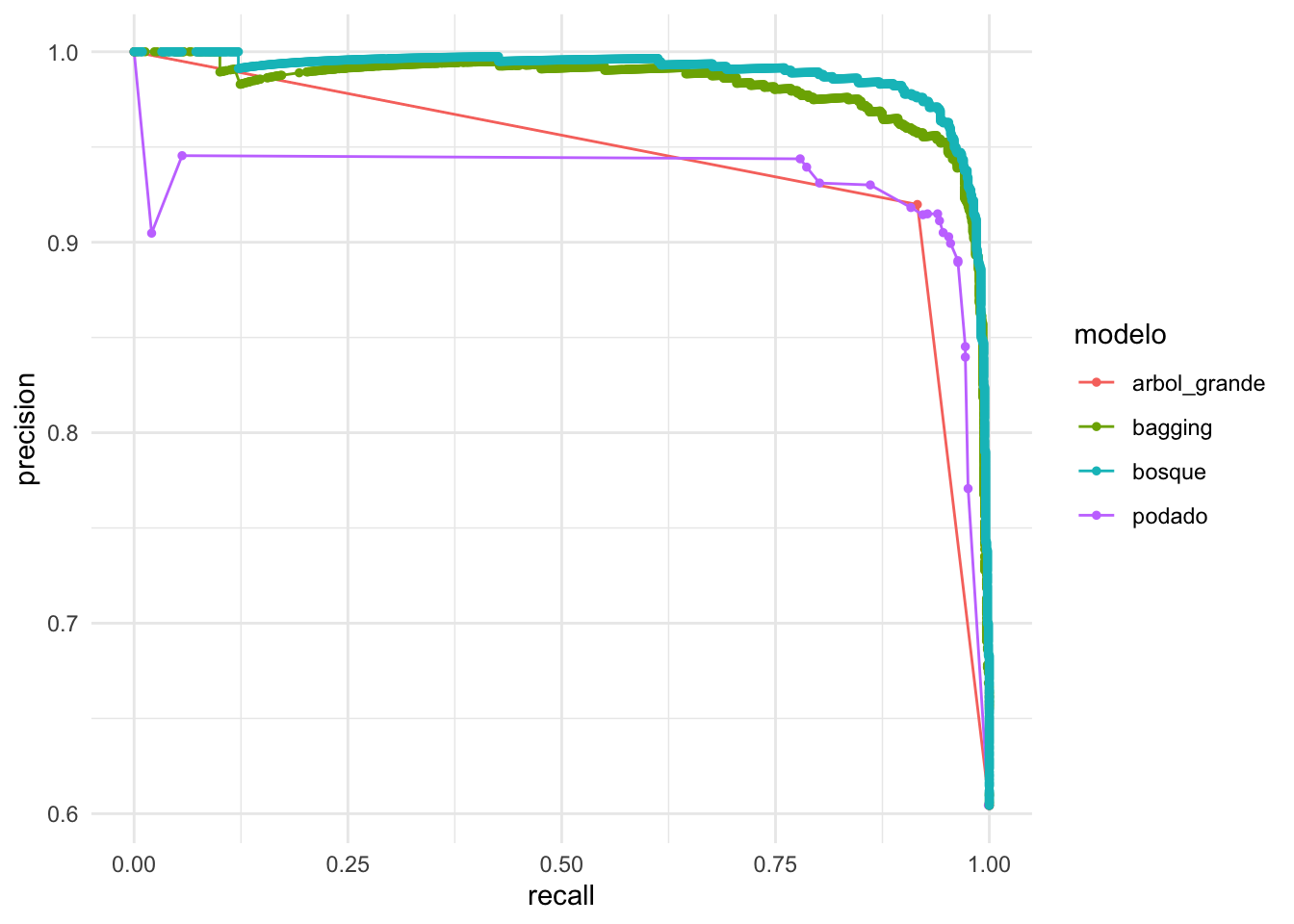
O las curvas ROC
roc_tbl <- map(names(modelos), function(mod_nombre){
predict(modelos[[mod_nombre]], spam_prueba, type = "prob") |>
bind_cols(spam_prueba |> select(spam)) |>
roc_curve(spam, .pred_no_spam) |>
mutate(modelo = mod_nombre)
}) |>
bind_rows()
ggplot(roc_tbl,
aes(x = 1 - specificity, y = sensitivity, colour = modelo)) +
geom_point(size= 0.5) + geom_path()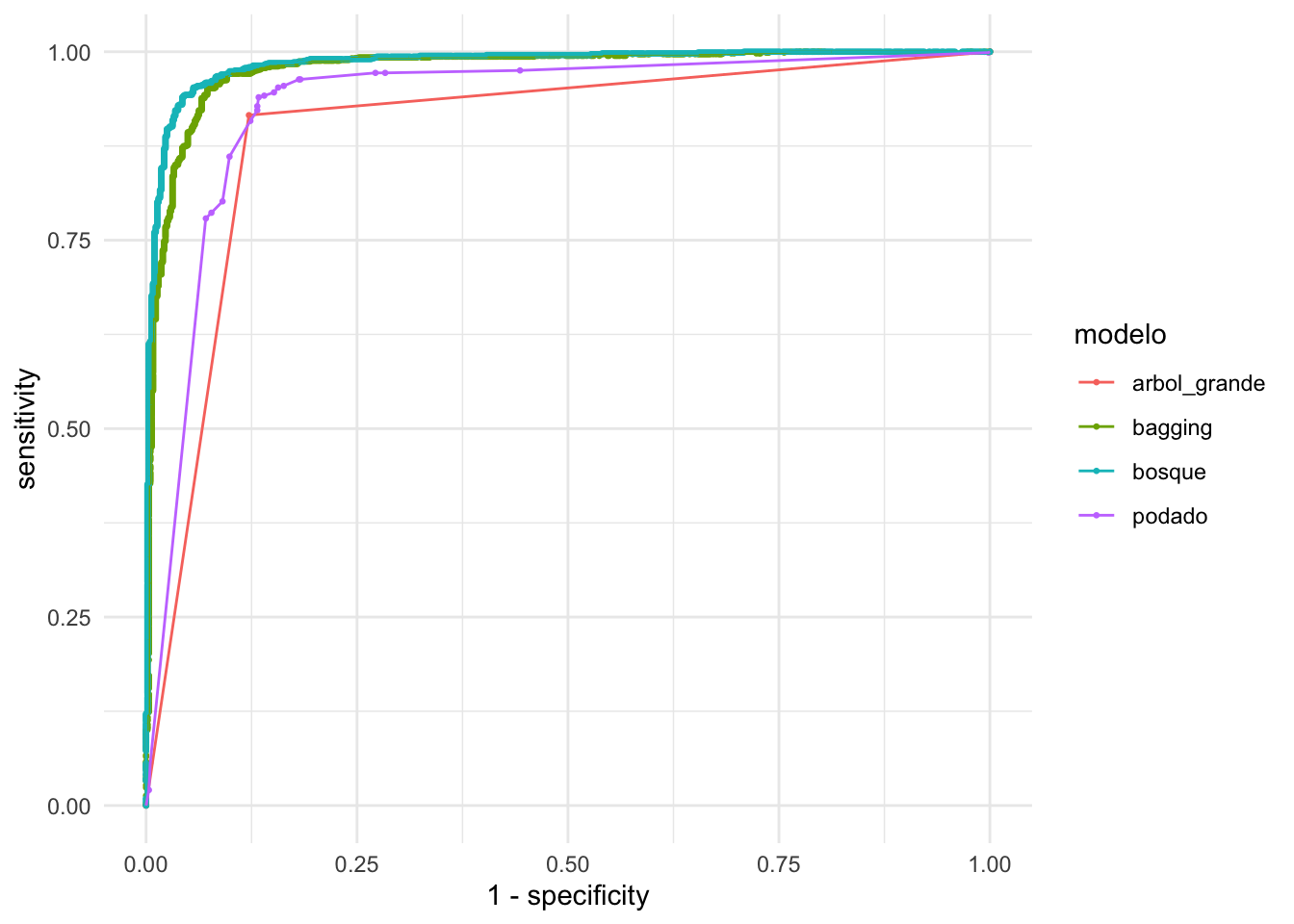
9.5 Ajustando árboles aleatorios.
- El parámetro más importante de afinar es usualmente \(m\), el número de variables que se escogen al azar en cada nodo.
- A veces podemos obtener algunas ventajas de afinar el número mínimo de observaciones por nodo terminal y/o el número mínimo de observaciones por nodo para considerar hacer cortes adicionales
- Usualmente corremos tantos árboles como podamos (cientos, miles), o hasta que se estabiliza el error. Aumentar más arboles rara vez producen sobreajuste adicional (aunque esto no quiere decir que los bosques aleatorios no puedan sobreajustar)
Implementaciones: hay distintas implementaciones con diferencias considerables. En nuestros ejemplos usamos el paquete ranger. En esta implementación, por ejemplo, las variables cualitativas se toman como ordenadas (alfabético si no es un factor, en el orden de los factores o, ordenadas según la variable respuesta si se usa la opción respect.unordered.factors = TRUE), ver documentación y referencias asociadas. Se puede usar esta última opción y el bosque resultante no necesariamente agrupa niveles de la variable.
9.6 Ventajas y desventajas de bosques aleatorios
Ventajas:
- Entre los métodos estándar, es en general uno de los métodos más competitivos: usualmente tienen tasas muy buenas de error de predicción.
- Los bosques aleatorios son relativamente fáciles de entrenar (ajustar usualmente 1 o 2 parámetros) y rápidos de ajustar.
- Heredan las ventajas de los árboles: no hay necesidad de transformar variables o construir interacciones (pues los árboles pueden descubrirlas en parte), son robustos a valores atípicos en las variables de entrada.
- Igual que con los árboles, las predicciones de los bosques siempre están en el rango de las variables de predicción (no extrapolan)
Desventajas: - Pueden ser lentos en la predicción, pues muchas veces requieren evaluar grandes cantidades de árboles. - No es tan simple adaptarlos a distintos tipos de problemas (por ejemplo, como redes neuronales, que combinando capas podemos construir modelos ad-hoc a problemas particulares). - La falta de extrapolación puede ser también un defecto (por ejemplo, cuando una estructura lineal aproximada es apropiadas).